Average Nucleotide Identity (ANI) 全称Average nucleotide identity,即平均核苷酸多态性,主要用于在全基因组水平间评估物种间的亲缘关系,ANI值为95%被视为界定种的标准。
2018年1月1日起,微生物系统分类的国际权威《International Journal of Systematic and Evolutionary Microbiology》在其官网发布新闻“Genome sequencing data required with Taxonomic Deions”,国际系统与进化微生物学杂志(IJSEM)要求作者提供基因组测序数据,并在分类学描述中提供新的分类单元的描述。目前分类单元确定常用的方法主要有DNA-DNA杂交(简称DDH),16S rRNA鉴定以及ANI分析。DDH分析复杂耗时,16S rRNA 鉴定存在局限性(如某些物种的16S rRNA相似度高达99%,但DDH值却很低),因此ANI在当前具有更高的可信度。相同物种的基因组ANI大于95%,不同物种的基因组ANI小于95%,因此常以95%的ANI作为物种划分与物种聚类的标准。
JSpeciesWS提供在线的ANI分析,主要分析步骤如下:
1.获取基因组数据。主要来源于NCBI数据库(ftp://ftp.ncbi.nlm.nih.gov/genomes/genbank或者https://www.ncbi.nlm.nih.gov/genome)
JSpeciesWS提供基因组数据(http://jspecies.ribohost.com/jspeciesws/#genomesdb),可在网页中通过TCS检索后加入到比对序列中。
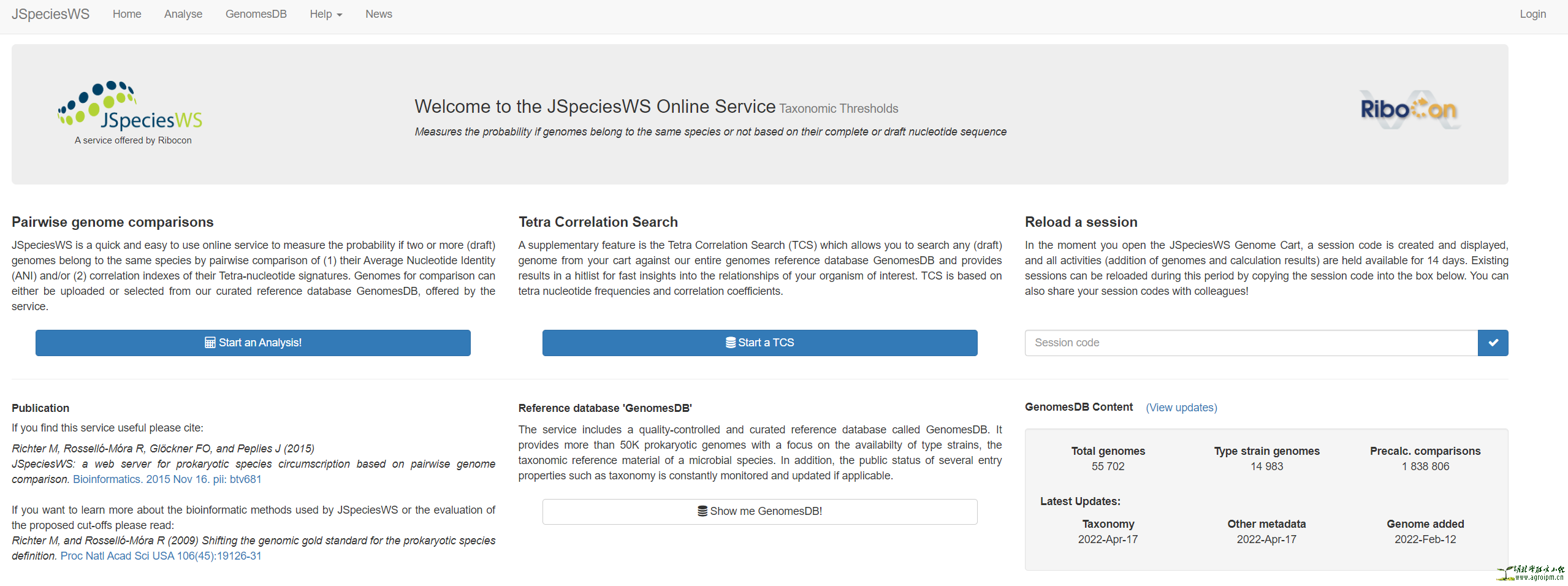
2.上传基因组数据。点击页面中的Start an Analysis (http://jspecies.ribohost.com/jspeciesws/#analyse),进入数据提交界面,将FASTA格式或经压缩成zip格式的压缩文件上传至系统。
3.开始分析。多种分析模式,可根据需要选择。填入邮箱,系统分析完成后将向邮箱发送信息,包括结果获取码(Session code)。

ANIm:使用 MUMmer (NUCmer) 比对序列
ANIb:使用 BLASTN+ to 比对序列的 1020nt 片段
TETRA:计算每个序列的四核苷酸的频率
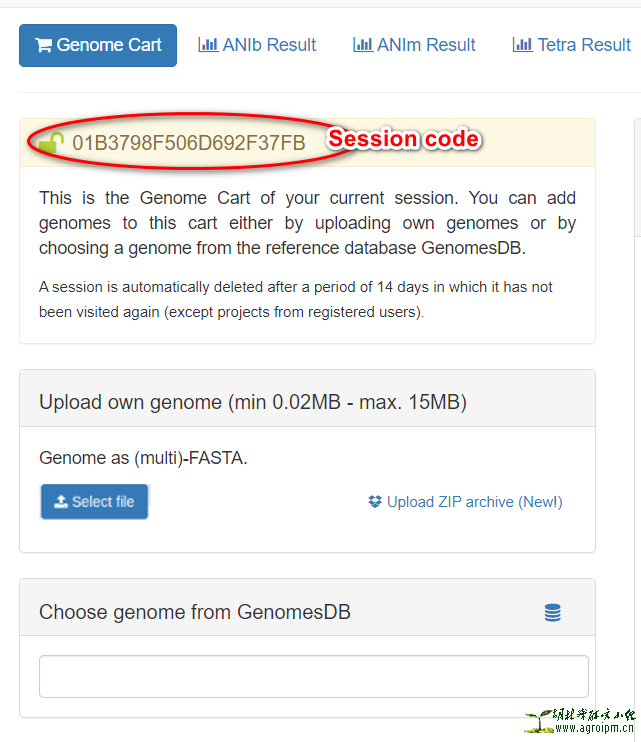
4.数据获取。每次分析产生一组会话代码(Session code),将该代码输入首页中Reload a session用于获取分析结果。结果可以csv格式下载保存。

同一种微生物在不同的国家或地区常有不同的名称,这就是俗名。俗名在局部地区可以使用,但不便于交流,容易引起混乱。为在世界范围...

以下内容转载自小木虫 哥特复兴VS [1]Han-Dong Sun,Yi-Ming Shi:LCUV-Guided Isolation and Structure Determination of Lancolide E: A Nortriterpenoid with a Tetracyclo [...

1 目的 规范TENSOR 27红外光谱仪的操作程序,正确使用仪器,保证设备安全和检测的顺利进行。 2 使用环境 电源电压:85~265V,47~65Hz 温度范...

人是能够思想的苇草 帕斯卡尔 人只不过是一根苇草,是自然界最脆弱的东西;但他是一根能思想的苇草。用不着整个宇宙都拿起武器来才能...